graphSAGE的论文,折腾GCN的人必读一篇论文。GCN计算需要完整图结构,既消耗硬件资源、也没法进行mini-batch学习。
本论文提出了graphSAGE模型,其核心思想是,通过对学习过程进行采样,近似完成GCN的计算过程,从而可以分批采样以达到进行batch学习的目的。另外对于图中新增的节点,只需要对新添加的节点进行采样学习即可。
(这篇论文在2020年1月左右阅读)
研究背景
GCN的一个问题在于,每次学习都需要将整个图送入显存/内存中,资源消耗巨大。另外使用整个图结构进行学习,导致了GCN的学习的固化,图中一旦新增节点,整个图的学习都需要重新进行。这两点对于大数据集和项目实际落地来说,是巨大的阻碍。
graphSAGE的原理
和GCN的论文一样,本咸鱼选择跳过论文中的数学证明,网上有大把比我分析的好的文章,想看数学原理建议阅读论文本体或者去看其他大手子们的文章。我这里只写一些个人理解。
GCN的计算细节
了解了GCN的工作模式之后会发现一个事实,每轮卷积中,每个节点只和自己周围的节点交换信息。如果是单层GCN,那么每个节点只能接受到自己一跳内邻居的信息。两层的话则是两跳(通过一跳邻居间接传播过来),如图所示:
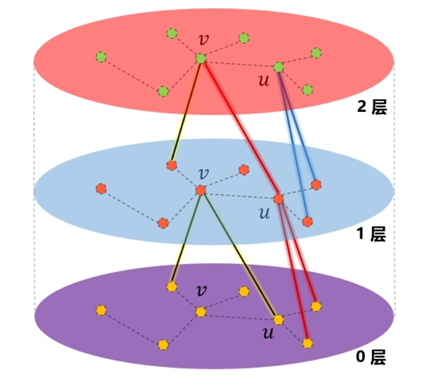
假设我只需要图中一个点的卷积的结果,并且只使用了一层GCN,那么实际上我只需要这个节点和它的全部邻居节点,图中的其他节点是没有意义的;
如果使用了两层GCN,那么我只需要这个节点和它的两跳以内的全部邻居,以此类推。因为更远节点的信息根本无法到达这个节点。
如此,便可以实现GCN的mini-batch,一次取出一组待训练节点和它们训练所需的N跳内的邻居节点即可,因为这些节点的训练本来也不需要更远的节点的信息。
通过采样进行近似
用过抽取出一部分待训练节点和它们的N跳内邻居可以完成这些节点的训练。但并不是说只进行这种操作就可以完成顺利mini-batch训练。例如,图中的某些节点是有几百上千个邻居的超级节点,对于这种节点,哪怕只进行一跳邻居采样,仍然会导致计算困难。
针对这个问题,本论文提出了采样的思路,即,只对目标节点的邻居进行一定数量的采样,然后通过这次被采样出来的节点和目标节点进行计算,从而逼近完全聚合的效果。
graphSAGE的具体实现
有了上述的分析,我们便清楚了graphSAGE的思路是,通过采样实现对整个图进行分批和近似计算。那么就有两个问题需要解决:如何进行采样以及如何进行计算。
采样的实现
作者没有在采样上折腾出花来,而是直接选择了随机采样。原版代码如下:
1 | class UniformNeighborSampler(Layer): |
可以看到是根据num_samples指定的数量,对邻居进行随机采样。
因为没有什么证据表明,邻居节点的分布遵循某个分布,所以普通的随机采样反而效果较稳定(另外作者可能试过按常见分布的采样方法,改几行代码的事),论文中有关于使用随机采样论证。
前面已经提过,由于GCN的特性:一层GCN中,节点只直接和邻居节点发生信息交换。所以叠加了几层GCN,就应该采样几跳内的邻居节点。
聚合特征的方法
采样完成后,等于是从原图中抽取出来了一个子图。对于这个batch的计算全部在这个子图上完成。
本论文并不是只实现了一个采样,然后计算部分全部照搬GCN(我导师管这种情况叫创新性不够)。而是自己提出了几个聚合信息的方法:
- 均值聚合:普通均值聚合、基于修正的均值聚合
- LSTM聚合
- 池化聚合:均值池化、最大值池化
其中普通均值聚合是简单的将邻居节点取均值,然后和待运算节点连接后,进行一次矩阵变换。
而基于修正的均值聚合的计算公式如下。
这个算法在聚合邻居节点的特征时就已经有了一定的权重,近似模拟GCN的计算过程。
而LSTM聚合是将邻居节点逐个输入LSTM中,因为作者认为LSTM(RNNs)比均值聚合有着更强的特征提取能力。但LSTM是适合于编码顺序数据的,因此每次使用LSTM聚合计算时需要进行一次随机打乱。
而池化聚合是考虑到了GCN的特性,GCN本身就是在平滑同类节点的嵌入结果,而使用均值或最大值池化,更有利于提取同类节点的特征。计算公式如下:
(作者声称,实验结果显示,均值池化和最大值池化没有显著的性能差距,故他们将重点放在了最大值池化上)
实验结果
通常我是不提及实验结果的,因为反正结果做的差也不会发上来。不过这篇论文的结果不是一水的往一个方向倒。所以我把实验结果弄上来。
本论文使用了三个数据集:
- Web of Science引文数据集。目标是将学术论文分类为不同的主题
- Reddit帖子。目标是将发帖人归为不同社区的成员
- 各种生物学蛋白质的功能分类蛋白质-蛋白质相互作用(PPI)图。目标是对未知的PPI图进行概括
实验结果如下:
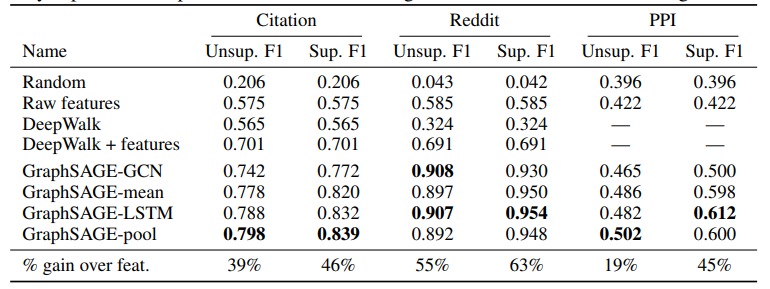
我想说的是:graphSAGE并没有duang的就在三个数据集上都表现出某一个聚合方法比较好。四个方法各有千秋,如果要用graphSAGE建议都试试。
总结
这篇论文也是2017年的了,后面又有一些其他七七八八的针对graph进行采样从而实现mini-batch的算法,某些方面比这篇文章更好,不过这篇文章仍然是GCN项目落地的必读文章之一。